Vivimos en un tiempo muy interesante en la historia humana. Con la explosión del desarrollo tecnológico en las últimas décadas, cada año más y más tecnologías que fueron consideradas ciencia ficción cobran vida, haciéndonos soñar lo que el futuro nos depara. Desde implantes oculares y brazos robóticos controlados con la mente, hasta la posibilidad de imprimir una casa, este año tuvimos avances muy interesantes.
- Implantes de ojos le dan visión a ciegos

Implante que permite a ciegos ver Dos hombres ciegos en el Reino Unido fueron provistos con implantes oculares durante una cirugía de ocho horas con resultados prometedores. Después de años de ceguera, ambos han recuperado visión “útil” en solo semanas, capturando los bordes de objetos y soñando en color. Los doctores esperan una mejora continua conforme sus cerebros se reconectan para la visión.Fuente: telegraph.co.uk
- La NASA comienza a utilizar exoesqueletos

Exoesqueleto de la NASA El Exoesqueleto robótico X1 pesa 23 kilos y contiene cuatro articulaciones motorizadas con seis pasivas. Con dos configuraciones, puede ya sea dificultar el movimiento, lo que le permitiría a los astronautas ejercitar en el espacio, o mejorar el movimiento, asistiendo a parapléjicos a caminar.Fuente: news.cnet.com
- Usando la mente para controlar un brazo robótico

Brazo robótico controlado por la mente En la Universidad de Pittsburgh, el departamento de neurobiología trabajó con Jan Scheuermann durante el transcurso de 13 semanas para crear un brazo robótico controlado solo por el poder de la mente de Scheuermann.El equipo implantó en ella dos microelectrodos intracorticales de 96 canales. Ubicados en la corteza motora, que controla todos los movimientos de extremidades, el proceso de integración fue más rápido de lo que todos esperaban. Al segundo día, Jan podía usar su nuevo brazo en un espacio tridimensional. Al final de las 13 semanas, era capaz de realizar tareas complejas con movimiento en siete dimensiones, igual que un brazo biológico.
Fuente: gizmodo.com
- Imprimiendo casas con una impresora tridimensional

Casa creada por impresora tridimensional La impresora en forma de “D”, creada por Enrico Dini, es capaz de imprimir un edificio de dos pisos, completo con cuartos, escaleras, tuberías y particiones. Usando nada más que arena y un compuesto orgánico para unirla, el material resultante tiene la misma durabilidad del concreto reforzado con una apariencia de mármol. El proceso de construcción se lleva aproximadamente un cuarto del tiempo de los edificios tradicionales, mientras que se ajuste a estructuras redondeadas, y puede ser construido sin conocimientos o habilidades especializadas.Fuente: gizmag.com
- El Bosón de Higgs fue encontrado

Bosón de Higgs encontrado Durante el verano, el centro de investigación multinacional CERN confirmó que había descubierto una partícula que se comportaba lo suficiente como lo esperado del Bosón de Higgs como para darle el nombre. Para los científicos, esto significa que puede haber un campo de Higgs, similar a un campo electromagnético. A su vez, esto podría darle a los científicos la habilidad de interactuar con la masa de la misma forma que interactuamos con los campos magnéticos.Fuente: forbes.com
- Piel en spray

Piel en Spray ReCell por Avista Medical es un gran avance médico para las víctimas de quemaduras severas. La tecnología utiliza una pieza de piel del tamaño de una estampilla del paciente, entonces la muestra es mezclada con una enzima cultivada de puercos y es aplicada de vuelta en el sitio quemado en forma de spray. Cada pequeño injerto se expande, cubriendo el espacio de una página de un libro dentro de una semana. Ya que la piel donada viene del mismo paciente, el riesgo de rechazo es mínimo.Fuente: news.discovery.com
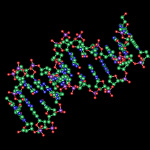
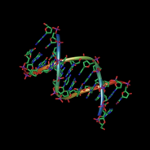
- ADN fotografiado por primera vez

Fotografía de ADN Usando un microscópio de electrones, Enzo di Fabrizio y su equipo en el Instituto Italiano de Tecnología en Genoa tomaron las primeras fotos de la famosa doble hélice.Fuente: newscientist.com
- Voyager I abandona el sistema solar

Voyager I Lanzado en 1977, el Voyager I es el primer objeto hecho por el hombre en volar más allá de los confines de nuestro sistema solar hacía la oscuridad del espacio profundo. Fue originalmente diseñado para enviar a casa imágenes de Saturno y Júpiter, pero los científicos de la NASA se dieron cuenta de que eventualmente la sonda podría flotar hacia el gran desconocido. Con ese propósito, una grabación fue colocada en el Voyager I con sonidos que van desde música hasta los llamados de ballenas, y saludos en 55 lenguajes.Fuente: space.com
- Quijada implantada fue creada con impresora tridimensional
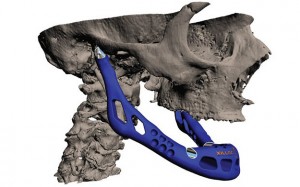
Quijada creada con impresora tridimensional Una quijada personalizada fue creada para un paciente de 83 años utilizando polvo de titanio y recubrimiento biocerámico. La exitosa cirugía, primera de este tipo, abre la puerta a reemplazos óseos personalizados y, quizá un día, la habilidad de imprimir nuevos músculos y órganos.Fuente: telegraph.co.uk
- El cerebro humano fue hackeado

Dispositivo que lee ondas cerebrales Usenix Security puso a un equipo de investigadores a que usaran tecnología ampliamente disponible para mostrar cuan vulnerable es realmente el cerebro humano. Con una diadema equipada para obtener electroencefalografías y el software para encontrar lo que las neuronas activándose están intentando hacer, busca picos en actividad cerebral cuando el usuario reconoce algo como un número del PIN de un cajero automático o la cara de un niño.Fuente: extremetech.com