Un nuevo método de interferencia de Ácido Ribonucleico (ARN, o RNA por sus siglas en inglés de RiboNucleic Acid) muestra promesa para tratar el cáncer, y otras enfermedades.
Anne Trafton, MIT News Office. Original (en inglés).
Durante la década pasada, científicos han estado siguiendo tratamientos de cáncer basados en interferencia de ARN – un fenómeto que ofrece una manera de apagar los genes con mal funcionamiento con pequeños trozos de ARN. Sin embargo, queda un enorme desafío: encontrar una manera de entregar eficientemente el ARN.
La mayoría del tiempo, ARN pequeño de interferencia (siRNA por sus siglas en inglés de small interfering RNA) – el tipo usado para interferencia ARN – es disuelto rápidamente dentro del cuerpo por enzimas que defienden contra infecciones por virus ARN.
“Ha sido una verdadera lucha el tratar de diseñar un sistema de entrega que nos permita administrar siRNA, especialmente si quieres apuntarle a partes específicas del cuerpo”, dijo Paula Hammond, Profesora de Ingeniería del David H. Koch en el MIT.
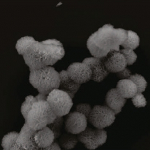
Hammond y sus colegas han llegado con un novedoso vehículo de entrega en el que el ARN es empacado dentro de microesferas tan densas que pueden resistir la degradación hasta alcanzar sus destinos. El nuevo sistema, descrito el 26 de febrero en el diario “Nature Materials”, derriba la expresión de genes específicos tan efectivamente como los métodos existentes de entrega, pero con una dosis mucho menor de partículas.
Dichas partículas podrían ofrecer una nueva manera de tratar no solo el cáncer, sino también cualquier otra enfermedad crónica causada por un “gen que no se comporta”, dijo Hammond, quien también es miembro del Instituto David H. Koch para Investigación de Cáncer Integrativa. “Interferencia de ARN tiene una enorme promesa para un gran número de enfermedades, una de las cuales es el cáncer, pero también enfermedades neurológicas y enfermedades inmunes”, dijo.
El autor líder de la revista académica es Jong Bum Lee, un antiguo postdoctorado en el laboratorio de Hammond. El postdoctorado Jinkee Hong, el doctor Daniel Bonner y el doctor Zhiyong Poon también son autores de la revista académica.
Interrupción genética
La interferencia de ARN es un proceso que ocurre naturalmente, descubierto en 1998, que permite a células ajustar precisamente su expresión genética. La información genética normalmente se carga del ADN en el núcleo a los ribosomas, estructuras celulares donde se forman las proteínas. siRNA se une al mensajero ARN que carga esta información genética, destruyendo instrucciones antes de que alcances al ribosoma.
Los científicos trabajan en muchas maneras para replicar artificialmente este proceso para apuntar a genes específicos, incluyendo empacar siRNA en nanopartículas hechas de lípidos (grasas) o materiales inorgánicos como el oro. Aunque muchas de éstas han mostrado algo de resultados, una desventaja es que es difícil cargar grandes cantidades de siRNA en estos cargueros, por que los cortos filamentos no se empacan ajustadamente.
Para superar esto, el equipo de Hammond decidió empacar el ARN como un largo filamento que se doblaría en una pequeña y compacta esfera. Los investigadores usaron un método para sintetizar ARN conocido como transcripción de círculo rotatorio para producir filamentos extremadamente largos de ARN hechos de una secuencia repetidora de 21 nucleoides. Esos segmentos están separados por una extensión más corta que es reconocida por la enzima Dicer, que corta el ARN cuando encuentra esa secuencia.
Conforme el filamento de ARN es sintetizado, se dobla en hojas que entonces se auto-ensamblan en una esfera muy densa similar a esponja. Hasta medio millón de copias de la misma secuencia de ARN pueden ser empacadas en una esfera con un diámetro de solo dos micrones. Una vez que la esferas se forman, los investigadores las empacan en una capa de polímero cargado positivamente, que induce a las esperas a empacarse aún más apretadas (hasta un diámetro de 200 nanómetros) y también las ayuda a entrar en las células.
Después de que las esferas entran a una célula, la enzima Dicer corta el ARN en lugares específicos, liberando las secuencias siRNA de 21 nucleótidos.
Peixuan Guo, director del Centro de Desarrollo de Nanomedicina NIH en la Universidad de Kentucky, dijo que el aspecto más emocionante del trabajo es el desarrollo de un método de auto-ensamblado para partículas de ARN. Guo, quien no fue parte del equipo de investigación, agrega que las partículas podrían ser más efectivas en entrar en las células si fueran encogidas a escalas aún más pequeñas, cercanas a los 50 nanómetros.
Apuntando a tumores
En la revista académica de “Nature Materials”, los investigadores probaron sus esferas programándolas para liberar secuencias de ARN que apagaran un gen que provoca que las células de tumores brillen en ratones. Encontraron que podían alcanzar el mismo nivel de derribo de sistemas de entrega de nanopartículas convencionales, pero utilizando hasta mil veces menos partículas.
Las microesponjas se acumulan en los sitios de tumores a través de un fenómeno comúnmente utilizado para entregar nanopartículas: Los vasos sanguíneos que rodean tumores tienen “filtraciones,” lo que significa que tienen pequeños poros a través de los cuales muy pequeñas partículas pueden colarse.
En estudios futuros, los investigadores planean diseñar microesferas recubiertas con polímeros que específicamente apunten a células de tumores u otras células de enfermedades. También trabajan en esferas que carguen ADN, para un potencial uso en terapia genética.
Reimpreso con permiso de MIT News.
Fuente
http://web.mit.edu/ (en inglés)