Una nanopartícula de ácido nucleico posee menos riesgo de efectos secundarios y ofrece mejor precisión al apuntarla.
Anne Trafton, MIT News Office. Original (en inglés).

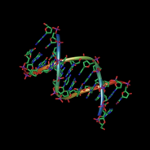
Usando una técnica conocida como “origami de ácido nucleico”, ingenieros químicos han construido pequeñas partículas hechas de ADN y ARN que pueden entregar trozos de ARN directamente a los tumores, apagando genes expresados en células de cáncer.
Para alcanzar este tipo de apagado de genes, conocido como interferencia ARN, mucho investigadores han tratado – con algo de éxito – de entregar ARN con partículas hechas de polímeros o lípidos. Sin embargo, esos materiales pueden poseer riesgos de seguridad y son difíciles de apuntar, dice Daniel Anderson, un profesor asociado de ciencias de la salud y tecnología e ingeniería química, y un miembro del Instituto David H. Koch para la Investigación Integrativa del Cáncer en el MIT (Massachusetts Institute of Technology – Instituto Tecnológico de Massachusetts).
Las nuevas partículas, desarrolladas por investigadores en el MIT, Alnylam Pharmaceuticals y la Escuela de Medicina de Harvard, parecen vencer aquellos desafíos, dice Anderson. Debido a que las partículas están hechas de ADN y ARN, son biodegradables y no poseen amenaza para el cuerpo. Pueden ser etiquetadas con moléculas de folato (la vitamina B9 o ácido fólico producida de manera natural por el cuerpo) para apuntar a la abundancia de receptores de folato encontrada en algunos tumores, incluyendo aquellos asociados con el cáncer de ovarios – uno de los cánceres más mortales y difíciles de tratar.
Anderson es autor principal de un artículo sobre las partículas que apareció en la edición del 3 de junio en Nature Nanotechnology. El autor líder del artículo es el antiguo posdoctorado del MIT Hyukjin Lee, ahora un profesor asistente en la Universidad de Mujeres Ewha en Seul, Corea del Sur.
Perturbación de genes
La interferencia ARN (RNAi por sus siglas en inglés), un fenómeno natural que las células usan para controlar su expresión genética, ha intrigado a los investigadores desde su descubrimiento en 1998. La información genética es normalmente cargada desde el ADN en el núcleo a ribosomas, estructuras celulares donde las proteínas son creadas. ARN interferente corto (siRNA por sus siglas en inglés de short interfering RNA), perturba este proceso al pegarse a las moléculas mensajeras ARN que cargan las instrucciones del ADN, destruyéndolas antes de que alcancen el ribosoma.
Nanopartículas que entregan siRNA hechas de lípidos, las que el laboratorio de Anderson y Alnylam también están desarrollando, han mostrado algo de éxito en apagar los genes del cáncer en estudios animales, y pruebas clínicas están ahora siendo llevadas a cabo en pacientes con cáncer de hígado. Las nanopartículas tienden a acumularse en el hígado, el bazo y los pulmones, así que el cáncer de hígado es un objetivo natural – pero ha sido difícil apuntar dichas partículas a tumores en otros órganos.
“Cuando piensas de cáncer metástatico, no quieres detenerte en el hígado”, dice Anderson. “También quieres llegar a más sitios diversos”.
Otro obstáculo para llenar la promesa del RNAi ha sido encontrar maneras de entregar las hebras cortas de ARN sin lastimar los tejidos saludables del cuerpo. Para evitar esos posibles efectos secundarios, Anderson y sus colegas decidieron entregar el ARN en un simple paquete hecho de ADN. Usando origami de ácido nucleico – que permite a los investigadores construir formas tridimensionales de segmentos cortos de ADN – fusionaron seis hebras de ADN para crear un tetraedro (una pirámide de seis bordes y cuatro caras). Una sola hebra de ARN fue entonces fijada a cada borde del tetraedro.
“Lo que es particularmente emocionante sobre el origami de ácido nucleico es el hecho de que puedes hacer partículas idénticas molecularmente y definir la localización de cada átomo”, dice Anderson.
Para apuntar las partículas a las células de tumor, los investigadores pegaron tres moléculas de folato a cada tetraedro. Los fragmentos de proteína cortos también podrían ser usados para apuntar las partículas a una variedad de tumores.
Usando origami de ácido nucleico, los investigadores tienen mucho más control sobre la composición de las partículas, volviendo más fácil crear partículas idénticas que todas busquen el mismo objetivo. Esto no es usualmente el caso con las nanopartículas de lípidos, dice Vinod Labhasetwar, un profesor de ingeniería biomédica en el Instituto de Investigación Lerner en la Clínica Cleveland. “Con partículas de lípidos, no estás seguro de qué fracción de las partículas realmente están llegando a los tejidos objetivo”, dice Labhasetwar, quien no estuvo involucrado en este estudio.
Circular y acumularse
En estudios de ratones implantados con tumores humanos, los investigadores encontraron que una vez inyectadas, las nanopartículas de ácido nucleico circularon en el torrente sanguíneo con una vida media de 24 minutos – el suficiente tiempo para alcanzar sus objetivos. El tetraedron de ADN parece proteger el ARN de la rápida absorción por los riñones y su excreción, lo que usualmente ocurre cuando el ARN es administrado por sí mismo, dice Anderson.
“Si tomas un ARN interferente corto y lo inyectas en el torrente sanguíneo, típicamente está fuera en seis minutos. Si haces una nanopartícula más grande usando métodos de origami, incrementa su habilidad para evitar la excreción a través de los riñones, incrementando por lo tanto su tiempo circulando por el corriente sanguíneo”, dice.
Los investigadores también mostraron que las nanopartículas de ácido nucleico se acumularon en los sitios del tumor. El ARN entregado por las partículas fue diseñado para apuntar a un gen por luciferasa (una enzima utilizada en bioluminiscencia), el cual fue agregado a las células del tumor para hacerlas brillar. Encontraron que en ratones tratados, la actividad de la luciferasa cayó más de la mitad.
El equipo diseña ahora nanopartículas para apuntar a genes que promueven el crecimiento del tumor, y también trabaja en apagar genes involucrados en otras enfermedades genéticas.
La investigación fue patrocinada por el Instituto Nacional de Salud (National Institutes of Health) de los Estados Unidos, el Centro para la Excelencia de la Nanotecnología del Cáncer (Cancer Nanotechnology Excellence), Alnylam Pharmaceuticals y la Fundación Nacional de Investigación de Corea.
Reimpreso con permiso de MIT News.
Fuente
http://web.mit.edu/ (en inglés)