Ingenieros diseñan nuevas proteínas que puedan ayudar a controlar los novedosos circuitos genéticos en las células.
Anne Trafton, MIT News Office. Original (en inglés).
Por cerca de 12 años, los biólogos sintéticos han estado trabajando en maneras de diseñar circuitos genéticos para realizar funciones novedosas como fabricar nuevas drogas, producir combustible e incluso programar el suicidio de células cancerosas.
Alcanzar estas complejas funciones requiere controlar muchos componentes genéticos y celulares, incluyendo no solo genes sino también las proteínas regulatorias que los encienden y los apagan. En una célula viviente, las proteínas llamadas factores de transcripción comúnmente regulan este proceso.
Hasta ahora, la mayoría de los investigadores han diseñado sus circuitos genéticos usando factores de transcripción encontrados en bacterias. Sin embargo, estos no siempre se traducen bien a células no bacteriales y puede ser un desafío aescalarlos, haciendo más difícil crear circuitos complejos, dice Timothy Lu, profesor asistente de ingeniería eléctrica y ciencia computacional y un miembro del Laboratorio de Investigación de Electrónica.
Lu y sus colegas en la Universidad de Boston, la Escuela de Medicina de Harvard y el Hospital General de Massachusetts han encontrado un nuevo método para diseñar factores de transcripción para células no bacteriales (en este caso, células de levadura). Su librería inicial de 19 factores de transcripción debería ayudar a superar el cuello de botella existente que ha limitado las aplicaciones de la biología sintética, dice Lu.
Este proyecto es parte de un esfuerzo más grande que se está llevando a cabo para desarrollar “partes” genéticas que pueden ser ensambladas en circuitos para alcanzar funciones específicas. A través de este esfuerzo, Lu y sus colegas esperan hacer más fácil el desarrollo de circuitos para hacer exactamente lo que quiere un investigador.
“Si observas el registro de partes, muchas de estas partes vienen de un revoltijo de organismos diferentes. Los juntas en tu organismo elegido y esperas que funcione,” dice Lu, el autor correspondiente de un artículo describiendo la nueva técnica de diseño de factor de transcripción en la edición del 3 de agosto del diario Cell.
Los autores principales del artículo incluyen a Ahmad Khalil, profesor asistente de ingeniería biomédica en la Universidad de Boston, LU, y el posdoctorado de la universidad de Boston Caleb Bashor. Otros autores son la estudiante graduada de Harvard Cherie Ramirez; la investigadora asistente de la Universidad de Boston Nora Pyenson; Keith Joung, jefe asociado de patología para la investigación en el Hospital General de Massachusetts; y James Collins, profesor de ingeniería biomédica en la Universidad de Boston.
Uniendo ADN
Avances recientes en el diseño de proteínas que unen el ADN le dieron a los investigadores el impulso que necesitaban para comenzar a contruir una nueva librería de factores de transcripción.
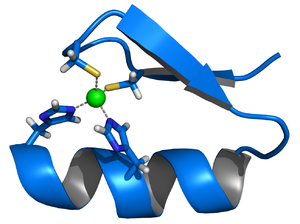
Los factores de transcripción incluyen una sección que reconoce y se anexa a una secuencia específica de ADN llamada promotor. La proteína recluta entonces una enzima llamada ARN polimerizado, que comienza el copiado del gen en el mensajero ARN, la molécula que carga las instrucciones genéticas al resto de la célula.
En muchos factores de transcripciones, la sección que une el ADN consiste de proteínas conocidas como dedos de cinc, que apuntan a diferentes secuencias de ADN dependiendo de su estructura. Los investigadores basaron sus nuevos diseños de dedos de cinc en la estructura de una proteína dedo de cinc que ocurre naturalmente. “Al modificar los aminoácidos específicos dentro del dedo de cinc, puedes hacer que se unan con nuevas secuencias objetivo”, dice Lu.
Los investigadores conectaron los nuevos dedos de cinc a segmentos activadores existentes, permitiéndoles crear muchas combinaciones de fuerza variable y especificidad. También diseñaron factores de transcripción que trabajan juntos, para que un gen solo pueda ser encendido si los factores se unen uno con el otro.
Andrew Ellington, un profesor de bioquímica en la Universidad de Texas en Austin, dice que el trabajo es un importante paso hacia crear circuitos más complejos en células no bacteriales. “Están creando un montón de nuevos factores de transcripción, y lo han hecho en una manera modular, creando herramientas adicionales que la gente puede usar para diseñar nuevos circuitos”, dice Ellington, quien no fue parte del equipo investigador.
Hacia mayor complejidad
Dichos factores de transcripción deberían hacer hacer más fácil para los biólogos sintéticos el diseñar circuitos para realizar tareas como sentir las condiciones ambientales de una célula.

En este artículo, los investigadores contruyeron algunos circuitos simples en levadura, pero planean desarrollar circuitos más complejos en estudios futuros. “No contruimos un circuito masivo de 10 o 15 factores de transición, pero eso es algo que definitivamente estamos planeando hacer en el futuro”, dice Lu. “Queremos ver que tanto podemos escalar el tipo de circuitos que podemos construir con este marco de trabajo”.
Los circuitos de biología sintética pueden ser análogos o digitales, al igual que los circuitos eléctricos. Los circuitos digitales incluyen funciones lógicas como compuertas AND y OR, que le permiten a las células hacer decisiones inequívocas como si deben pasar por un suicidio celular programado. Las funciones análogas son útiles para sensores que toman mediciones continuas de una molécula específica en la célula o su entorno. Al combinar estos circuitos, los investigadores pueden crear sistemas más complejos en los que una decisión digital sea activada una vez que el sensor alcanza un cierto umbral.
Además de construir circuitos más complejos, los investigadores están planeando tratar sus nuevos factores de transcripción en otras especies de levadura, y eventualmente en células de mamíferos, incluyendo células humanas. “Lo que realmente esperamos al final del día es que la levadura sea una buena plataforma de lanzamiento para diseñar estos circuitos”, dice Lu. “Trabajando en células de mamíferos es más lento y tedioso, así que si podemos construir circuitos verificados y partes en levadura y entonces importarlos, eso sería la situación ideal. Pero no hemos probado que podemos hacer eso todavía”.
La investigación fue patrocinada por el Instituto Médico Howard Hughes, los Institutos Nacionales de Salud, la Oficina de Investigación Naval, la Agencia de Proyectos de Investigación Avanzada para la Defensa (DARPA – Defense Advanced Research Projects Agency) y la Fundación Nacional de Ciencia, todos de los Estados Unidos.
Reimpreso con permiso de MIT News.
Fuente
http://web.mit.edu/ (en inglés)