Un nuevo estudio del MIT (Massachusetts Institute of Technology – Instituto Tecnológico de Massachusetts) ofrece una mirada profunda a los cambios químicos y genéticos que ocurren cuando la inflamación progresa en cáncer.
Anne Trafton, MIT News Office. Original (en inglés).
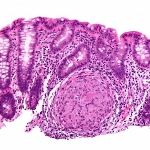
Imagen: wikipedia/nephron
Uno de los factores de riesgo más grandes para el cáncer de hígado, colon o estómago es la inflamación crónica de esos órganos, comúnmente causada por infecciones virales o bacteriales. Un nuevo estudio del MIT ofrece la mirada más profunda hasta ahora sobre cómo dichas infecciones provocan que los tejidos se vuelvan cancerosos.
El estudio, que apareció en la edición en línea de Proceedings of the National Academy of Sciences (PNAS) de la semana del 11 de Junio, rastreó una variedad de cambios genéticos y químicos en los hígados y cólones de ratones infectados con Heliobacter hepaticus, una bacteria similar a la Helicobacter pylori, la que causa úlceras estomacales y cáncer en humanos.
Los hallazgos podrían ayudar a los investigadores a desarrollar maneras de predecir las consecuencias a la salud de inflamación crónica, y diseñar drogas que detengan dicha inflamación.
“Si entiendes el mecanismo, entonces puedes diseñar intervenciones”, dice Peter Dedon, un profesor de Ingeniería Biológica del MIT. “Por ejemplo, ¿qué tal si desarrollamos maneras de bloquear o interrumpir los efectos tóxicos de la inflamación crónica?”.
Dedon es uno de los autores principales del artículo, junto con Steven Tannenbaum, un profesor de ingeniería biológica y química; James Fox, un profesor de ingeniería biológica y director del Departamento de Medicina Comparativa; y Gerald Wogan, un profesor de ingeniería biológica y química. El autor principal es Aswin Mangerich, un antiguo posdoctorado del MIT ahora en la Universidad de Konstanz en Alemania.
Demasiado de una cosa buena
Durante los últimos 30 años, Tannenbaum ha liderado un grupo de investigadores del MIT dedicados a estudiar el vínculo entre inflamación crónica y cáncer. La inflamación es una de las reacciones naturales del cuerpo a cualquier tipo de infección o daño, pero cuando se prolonga por mucho tiempo, los tejidos pueden ser dañados.
Cuando el sistema inmune del cuerpo detecta patógenos o daño celular, activa un torrente de células llamadas macrófagos y neutrófilos. El trabajo de estas células es devorar bacterias, células muertas y escombros: proteínas, ácidos nucleicos y otras moléculas liberadas por células muertas o dañadas. Como parte de este proceso, las células producen químicos altamente reactivos que ayudan a degradar a las bacterias.
“Al hacer esto, devorar las bacterias y soltar estos químicos reactivos sobre ellas, los químicos también pueden difundirse en el tejido, y ahí es donde se presenta el problema”, dice Dedon.
Si esto se lleva a cabo durante un largo período de tiempo, esa inflamación puede eventualmente llevar al cáncer. Un estudio reciente publicado en el diario The Lancet encontró que las infecciones cuentan por alrededor de 16 por ciento de nuevos casos de cáncer en el mundo.
Daño extendido
En el nuevo estudio del MIT, los investigadores analizaron ratones que habían sido infectados con H. hepaicus, que causa que desarrollen una condición similar a la enfermedad inflamatoria intestinal en los humanos. Durante el transcurso de 20 semanas, los ratones desarrollaron infecciones crónicas del hígado y el colon, con algunos de los ratones desarrollando cáncer de colon.
A lo largo del período de 20 semanas, los investigadores midieron alrededor de una docena de tipos diferentes de daño al ADN, ARN y las proteínas. También examinaron el daño al tejido y midieron que genes fueron encendidos y apagados según la infección progresó. Uno de sus encuentros claves fue que el hígado y el colon respondieron diferente a la infección.
En el colon, pero no en el hígado, el neutrófilo secretó ácido hipocloroso (también encontrado en los limpiadores caseros), que daña significativamente las proteínas, el ADN y el ARN añadiendo un átomo de cloro a ellos. El ácido hipocloroso tiene la intención de matar a las bacterias, pero también puede filtrarse en el tejido circundante y daña las células epiteliales del colon.
Los investigadores encontraron niveles de uno de los productos del daño por cloro en el ADN y ARN, clorotirosina, bien correlacionada con la severidad de la inflamación, lo que podría permitirles predecir el riesgo de inflamación crónica en pacientes con infecciones del colon, hígado o estómago. Tannenbaum recientemente identificó otro producto del daño por cloro en proteínas: clorotirosina, la que se correlaciona con inflamación. Mientras que estos resultados apuntan a un papel importante de los neutrófilos en la inflamación y el cáncer, “aún no sabemos si podemos predecir el riesgo de cáncer de estas moléculas dañadas”, dice Dedon.
Otra diferencia que encontraron los investigadores entre el colon y el hígado fue que los sistemas de reparación de ADN se volvieron más activos en el hígado pero menos activos en el colon, aún cuando ambos estaban experimentando daño de ADN. “Es posible que tengamos un doble efecto [en el colon]. Tienes estas bacterias que suprimen la reparación de ADN, al mismo tiempo que tienes todo este daño al ADN ocurriendo en el tejido como resultado de la respuesta inmune a las bacterias”, dice Dedon.
Los investigadores también identificaron varios tipos de daño al ADN previamente desconocidos en ratones y humanos, uno de los que involucran la oxidación de la guanina, un bloque de construcción de ADN, en dos nuevos productos, spiroiminodihidatoina (spiroiminodihydantoin) y guanidinohidanotoina (guanidinohydanotoin).
James Swenberg, un profesor de ciencias ambientales e ingeniería en la Universidad de la Escuela de Salud Pública de Carolina del Norte, dice que estudios “profundos e inovativos” deberían ayudar a los investigadores a entender mejor muchos tipos de cáncer. “No puedo recordar haber visto un artículo que trajo tantos aspectos de investigación a la mesa en un reporte”, dijo Swenberd, quien no estuvo involucrado en el estudio.
En futuros estudios, el equipo del MIT planea investigar los mecanismos del desarrollo del cáncer con más detalle, incluyendo ver por qué las células experimentan una disminución en algunos tipos de daño al ADN pero no en otros.
La investigación fue patrocinada por el Instituto Nacional del Cáncer de los Estados Unidos.
Reimpreso con permiso de MIT News.
Fuente
http://web.mit.edu/ (en inglés)