Los lípidos microbiales resultantes también podrían significar caídas de oxígeno en la historia geológica de la Tierra.
Jennifer Chu, MIT News Office. Original (en ingles)
En los días que siguieron el derrame petrolero del Deepwater Horizon, bacterias que consumen metano prosperaron en el Golfo de México, alimentándose del metano que brotó, junto con el petróleo, del pozo dañado. La súbita afluencia de microbios fue una curiosidad científica: Anteriormente al derrame petrolero, científicos habían observado relativamente pocos signos de microbios que consumían metano en el área.
Ahora investigadores del MIT han descubierto un gen bacterial que podría explicar esta súbita afluencia de bacterias que se alimentan de metano. Este gen le permite a las bacterias sobrevivir en entornos extremos y carentes de oxígeno, durmientes hasta que la comida – como el metano de un derrame petrolero, y el oxígeno necesario para metabolizarlo – se vuelve disponibles. El gen codifica una proteína, llamada HpnR, que es responsable por producir lípidos bacteriales conocidos como 3-metilhopanoides. Los investigadores dicen que producir estos lípidos podría preparar mejor a los microbios para hacer una aparición súbita en la naturaleza cuando las condiciones son favorables, como después del accidente del Deepwater Horizon.
Los lípidos producidos por la proteína HpnR también podrían ser usados como biomarcadores, o una firma en las capas de roca, para identificar cambios dramáticos en los niveles de oxígeno en el transcurso de la historia geológica.
“Lo que nos interesa es que esto podría ser una ventana al pasado geológico”, dice la posdoctorado Paula Welander del Departamento de Ciencias de la Tierra, Atmosféricas y Planetarias (EAPS – Earth, Atmospheric and Planetary Sciences) del MIT (Massachusetts Institute of Technology – Instituto Tecnológico de Massachusetts). “En el registro geológico, hace muchos millones de años, vemos un número de eventos de extinciones masivas donde hay evidencia de agotamiento de oxígeno en el océano. Es en estos eventos clave, e inmediatamente después de estos, donde también vemos un incremento en todos los biomarcadores como indicadores de una perturbación climática. Parece ser parte de un síndrome de calentamiento, deoxigenización del océano y extinción biótica. Las causas son desconocidas”.
Welander y el profesor de EAPS Roger Summons han publicado sus resultados esta semana en el Proceedings of the National Academy of Sciences (PNAS).
Una señal en las rocas
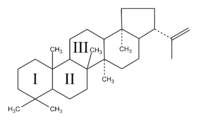
Las capas rocosas de la tierra sostienen restos de la evolución de la vida, desde las antiguas trazas de organismos unicelulares a los recientes fósiles de vertebrados. Uno de los biomarcadores clave que los geólogos han usado para identificar las formas tempranas de vida es una clase de lípidos llamados hopanoides, cuya robusta estructura molecular los ha preservado en el sedimento por miles de millones de años. Los hopanoides también han sido identificados en las bacterias modernas, y los geólogos estudiando los lípidos en las rocas antiguas los han usado como señales de la presencia de bacterias similares hace miles de millones de años.
Pero Welander dice que los hopanoides podrían ser usados para identificar más que las tempranas formas de vida: Los fósiles moleculares podrían ser biomarcadores para fenómenos ambientales – como períodos de muy bajo oxígeno.
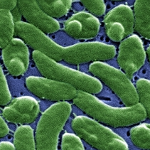
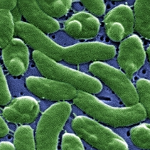
Para probar la teoría, Welander examinó una cepa moderna de la bacteria llamada Methylococcus capsulatus, un organismo ampliamente estudiado aislado por primera vez de un baño público romano antiguo en Bath, Inglaterra. El organismo, que también vive en entornos pobres en oxígeno como las ventosas en lo profundo del océano y los volcanes de lodo, ha sido de interés para los científicos por su habilidad de consumir eficientemente grandes cantidades de metano – lo que podría hacerla útil en biomediación y desarrollo de biocombustibles.
Para Welander y Summons, M. capsulatus es especialmente interesante por su estructura: El organismo contiene un tipo de hopanoide con una estructura molecular de cinco anillos que contiene metilación C-3. Los geólogos han encontrado que dichas metilaciones en la estructura de anillo son particularmente bien preservadas en rocas antiguas, aún cuando el resto del organismo ha desaparecido.
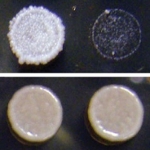
Welander estudió el genoma de la bacteria e identificó hpnR, el gen que codifica la proteína HpnR, el que está específicamente asociado con la metilación C-3. Entonces diseñó un método para borrar el gen, creando una cepa mutante. Welander y Summons entonces crecieron cultivos de cepas mutantes así como cultivos de bacterias salvajes (sin alteraciones). El equipo expuso ambas cepas a los niveles bajos de oxígeno y los altos niveles de metano durante un período de dos semanas para simular un entorno pobre en oxígeno.
Durante la primera semana, había poca diferencia entre los dos grupos, ambos de los cuales consumieron metano y crecieron a alrededor de la misma taza. Sin embargo, en el día 14, los investigadores observaron que la cepa salvaje comenzó a crecer más rápido que la bacteria mutante. Cuando Welander añadió el gen hpnR de vuelta en la bacteria mutante, encontró que eventualmente esta regresaba a los niveles que se asemejaban al de la cepa salvaje.
Apenas logrando sobrevivir
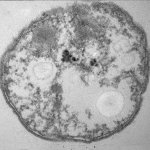
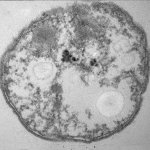
¿Qué podría explicar el contraste dramático en las tasas de sobrevivencia? Para responder esto, el equipo usó microscopía electrónica para examinar las estructuras celulares en las bacterias mutantes y salvajes. Descubrieron la marcada diferencia: Mientras que el tipo salvaje estaba lleno con membranas normales y vacuolas, la cepa mutante no tenía ninguna.
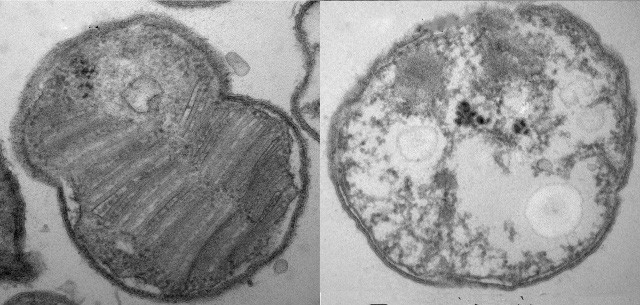
Las membranas faltantes, dice Welander, son una pista a la función del lípido. Ella y Summons postulan que el gen hpnR podría preservar las membranas celulares de las bacterias, lo que podría reforzar al microbio en tiempos de nutrientes agotados.
“Tienes a estas comunidades que apenas salen del paso, sobreviviendo en lo que pueden”, dice Welander. “Entonces cuando reciben una ráfaga de oxígeno o metano, pueden tomarlo muy rápidamente. Están realmente preparadas para aprovechar algo como esto”.
Los resultados, dice Welander, son especialmente emocionantes desde una perspectiva geológica. Si los 3-metilhopanoides realmente permiten a las bacterias sobrevivir en tiempos de oxígeno bajo, entonces un pico en el lípido relacionado en el registro rocoso podría indicar una disminución dramática en la historia de la Tierra, permitiendo a los geólogos entender mejor los períodos de extinciones masivas o grandes muertes masivas oceánicas.
“La meta original fue hacer esto un mejor biomarcador para los geólogos”, dice Welander. “Es [un trabajo] muy meticuloso, pero al final también queremos causar un mayor impacto, por ejemplo aprender como los microorganismos lidian con los hidrocarbonos en el entorno”.
David Valentine, un profesor de geoquímica microbial en la Universidad de California en Santa Bárbara, dice que el lípido objetivo del grupo es parecido al colesterol, que juega un papel importante en las membranas de células humanas y animales. Dice que el gen identificado por el grupo podría jugar un papel similar en bacterias.
“Este trabajo demuestra una importante unidad en biología”, dice Valentine. “Sus resultados son un paso necesario en proveer contexto para interpretar la distribución de estos biomarcadores en el registro geológico”.
Esta investigación fue patrocinada por la NASA y la Fundación Nacional de Ciencia de los Estados Unidos.
Reimpreso con permiso de MIT News.
Fuente
http://web.mit.edu/ (en inglés)